Wymagania
Program dostępny jest w wersjach przeznaczonych dla systemów operacyjnych Windows, Linux i MacOS X.
Niezależnie od systemu operacyjnego do działania programu konieczna jest obecność na komputerze maszyny wirtualnej Javy (JRE - Java Runtime Engine) w wersji 1.8 lub nowszej.
O tym, czy JRE w odpowiedniej wersji jest obecny w systemie można przekonać się wpisując z linii komend polecenie:
java -version
Jeżeli JRE jest zainstalowany, to efektem polecenia powinien być komunikat podobny do widocznego na poniższym obrazku.
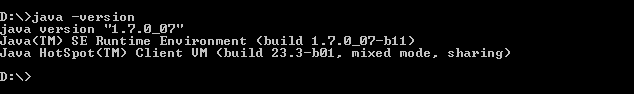
W przypadku braku JRE w odpowiedniej wersji konieczne jest jego ściągnięcie i instalacja. Najnowsze wersje JRE dla systemów Windows i Linux i macOS dostępne są na stronie firmy Oracle.
Instalacja
Instalacja w systemach Windows i Linux polega na rozpakowaniu zawartości archiwum z programem (odpowiednio MScan.zip lub MScan.tgz) do dowolnego katalogu.
W przypadku systemu MacOS X należy zamontować obraz dysku MScan.dmg i przegrać jego zawartość do dowolnego katalogu.
Uruchomienie
Pod systemami Windows i Linux program może być uruchomiony za pomocą dwukrotnego kliknięcia na ikony skryptów startowych (odpowiednio MScan.bat lub MScan.sh). Możliwe jest również uruchomienie skryptów z poziomu linii komend.
W systemie MacOS X program uruchamia się przez dwukrotne kliknięcie na ikonę MScan.app.